一、引言
乳腺癌是女性中最常见的恶性肿瘤之一,也影响着全球范围内许多人们的健康。据世界卫生组织(WHO)的数据,乳腺癌是全球癌症发病率和死亡率最高的肿瘤之一,其对个体和社会的危害不可忽视。因此,早期乳腺癌的预测和诊断变得至关重要,以便及早采取适当的治疗措施,提高治愈率和生存率。
为了提高乳腺癌预测的准确性和成功率,研究人员将基于主成分分析(PCA)和逻辑回归的方法应用于乳腺癌预测研究中。PCA作为一种降维技术,可以从众多特征中提取主要信息,并减少冗余特征的影响。逻辑回归则是一种常见的分类算法,通过建立一个预测模型来评估特征与乳腺癌之间的关系。这种组合方法可以在乳腺癌预测中起到关键作用,提高预测的准确性和可靠性。
本文旨在探讨如何利用PCA和逻辑回归方法来提高乳腺癌的预测成功率。通过主成分分析降维和逻辑回归分类模型的应用,可以有效地处理乳腺癌预测中复杂的特征数据,并提高预测的准确性和可靠性。这对于乳腺癌的早期诊断和治疗具有重要的临床意义,也为未来深入研究乳腺癌预测提供了一定的参考价值。
二、PCA(主成分分析)简介
2.1 PCA的基本原理和作用
主成分分析(Principal Component Analysis,简称PCA)是一种常用的数据分析方法,用于降低数据的维度。其基本原理是通过线性变换将原始数据转换为一组新的变量,称为主成分,这些主成分能够尽可能地保留原始数据的信息。每个主成分都是原始变量的线性组合,且彼此之间是相互独立的。
主成分分析(PCA)常用于处理「连续变量的数据」。PCA最适用于连续型变量,也就是数值型的数据,如测量结果、生物标志物、临床指标等。对于连续变量,PCA可以计算各个主成分的方差贡献率,并识别出数据中的相关结构和模式。
然而,如果数据中既包含连续变量又包含分类或有序变量,可以考虑使用其他方法,如多元方差分析(MANOVA)或偏最小二乘回归(PLSR),这些方法可以同时考虑不同类型的变量。「PCA的作用主要有两个方面」:
- 降维:PCA能够将原始高维数据转换为低维表示,减少特征的数量。通过选择保留的主成分数量,可以选择性地削减数据的维度,从而减少计算复杂度和存储空间的需求。
- 特征提取:PCA通过寻找数据中的主要信息,识别出与变量之间的相关性最大的主成分。这些主成分通常对数据的变异程度贡献最大,在数据分析和模型构建中具有重要的意义。
2.2 PCA在数据维度削减中的应用优势
- 去除冗余特征:通过PCA,我们可以通过保留能够解释大部分数据方差的主成分,去除与乳腺癌预测无关或冗余的特征。这样可以更好地集中于那些真正对乳腺癌预测有贡献的特征。
- 解决多重共线性问题:多重共线性是指特征之间存在高度相关性的情况,这会导致模型的不稳定性和低解释度。通过应用PCA,我们可以将高度相关的特征合并为一个主成分,从而减少共线性的影响,提高预测模型的可靠性。
- 可视化数据:由于PCA将高维数据转换为低维表示,我们可以将数据在二维或三维空间中进行可视化展示。这样可以更直观地观察数据的分布情况,有助于理解数据的结构和变异程度。
2.3 PCA为何适用于乳腺癌预测问题?
- 多个特征之间存在相关性:乳腺癌预测通常涉及多个特征,如乳房肿块、乳头溢液等。这些特征之间可能存在相关性,而PCA可以通过提取主成分来捕捉特征之间的相关性,从而减少数据的维度并保留最有信息量的特征。
- 数据维度较高:乳腺癌预测所使用的数据集通常包含大量特征,而高维数据会带来计算和存储上的挑战。应用PCA可以减少数据的维度,简化问题,并提高模型的训练和预测效率。
- 需要强调重要特征:乳腺癌预测中,某些特征可能对预测结果更为重要。通过PCA,我们可以选择保留那些解释数据变异最多的主成分,这样可以更加集中于那些对乳腺癌预测有关联的特征,提高预测的准确性。
总结:PCA通过降维和特征提取的方式,在乳腺癌预测问题中具有重要的应用优势。它能够削减数据维度、去除冗余特征、解决多重共线性问题,并突出重要特征。因此,PCA是一种适用于乳腺癌预测问题的有效方法。
三、PCA如何应用于乳腺癌预测
3.1 如何将PCA引入乳腺癌预测模型?
- 数据准备:收集和整理乳腺癌预测所需的特征数据,确保数据已经进行了预处理(如缺失值填充、标准化等)。
- PCA模型训练:使用原始特征数据训练PCA模型。在训练过程中,PCA会计算主成分的方差和协方差矩阵,并确定每个主成分的权重系数。
- 主成分选择:根据方差解释率或其他准则,选择保留的主成分数量。通常选择保留能够解释大部分数据方差(如80%以上)的主成分。
- 特征变换:将原始特征数据通过PCA模型进行转换,得到降维后的特征数据。这些降维后的特征即为选取的主成分。
3.2 如何通过PCA进行数据降维,提取关键特征?
- 计算协方差矩阵:对原始特征数据进行协方差矩阵的计算。协方差矩阵反映了特征之间的相关性。
- 特征值分解:对协方差矩阵进行特征值分解,得到特征值和对应的特征向量。
- 特征选择:根据特征值排序,选择保留的主成分数量。通常选择保留能够解释大部分数据方差的主成分。
- 特征变换:将原始特征数据通过选取的主成分进行线性变换,得到降维后的特征数据。
3.3 PCA在减少冗余信息和消除噪声有哪些作用?
- 冗余信息减少:PCA通过将高度相关的特征合并为较少数量的主成分,从而减少了数据中的冗余信息。保留的主成分尽量包含了原始数据中的大部分变异程度,以此来更好地代表原始数据集。
- 噪声消除:通过选择保留的主成分数量,PCA会筛选掉与预测目标不相关的特征,即那些对数据变异贡献较小的特征。这样可以减少噪声的影响,提高模型的鲁棒性和泛化能力。
- 数据压缩:PCA通过降低数据的维度,将原始数据转换为更紧凑的表示形式,从而实现数据压缩的效果。这不仅节省了存储空间,还减少了计算复杂度。
综上所述,PCA通过数据降维和特征提取的方式,减少了冗余信息和噪声的影响,使得乳腺癌预测模型更加简洁、高效和鲁棒。
四、示例与代码实现
- 「数据集准备」
library(survival) head(gbsg)
结果展示:
pid age meno size grade nodes pgr er hormon rfstime status 1 132 49 0 18 2 2 0 0 0 1838 0 2 1575 55 1 20 3 16 0 0 0 403 1 3 1140 56 1 40 3 3 0 0 0 1603 0 4 769 45 0 25 3 1 0 4 0 177 0 5 130 65 1 30 2 5 0 36 1 1855 0 6 1642 48 0 52 2 11 0 0 0 842 1
- 「示例数据集介绍」
> str(gbsg) 'data.frame': 686 obs. of 10 variables: $ age : int 49 55 56 45 65 48 48 37 67 45 ... $ meno : int 0 1 1 0 1 0 0 0 1 0 ... $ size : int 18 20 40 25 30 52 21 20 20 30 ... $ grade : int 2 3 3 3 2 2 3 2 2 2 ... $ nodes : int 2 16 3 1 5 11 8 9 1 1 ... $ pgr : int 0 0 0 0 0 0 0 0 0 0 ... $ er : int 0 0 0 4 36 0 0 0 0 0 ... $ hormon : int 0 0 0 0 1 0 0 1 1 0 ... $ rfstime: int 1838 403 1603 177 1855 842 293 42 564 1093 ... $ status : Factor w/ 2 levels "0","1": 1 2 1 1 1 2 2 1 2 2 ... age:患者年龄 meno:更年期状态(0表示未更年期,1表示已更年期) size:肿瘤大小 grade:肿瘤分级 nodes:受累淋巴结数量 pgr:孕激素受体表达水平 er:雌激素受体表达水平 hormon:激素治疗(0表示否,1表示是) rfstime:复发或死亡时间(以天为单位) status:事件状态(0表示被截尾,1表示事件发生)
- 「加载依赖库」
# 安装并加载所需的包 install.packages("factoextra") # 安装factoextra包 library(factoextra) # 加载factoextra包
- 「PCA主成分分析」
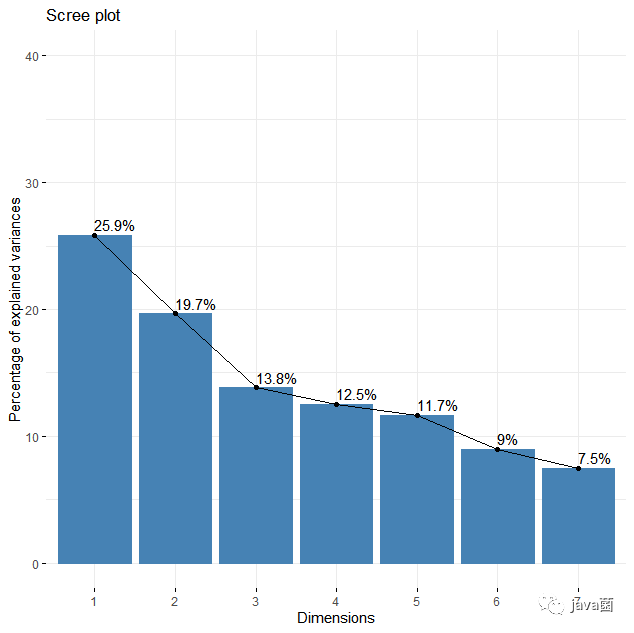
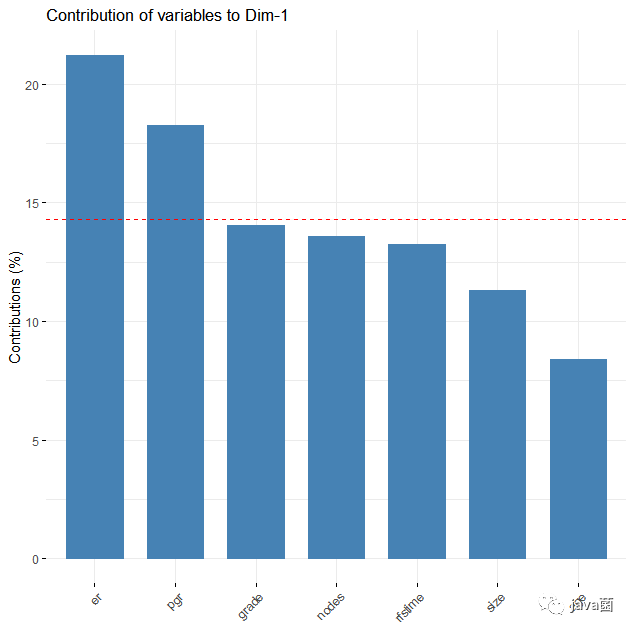
# 去除分类变量,PCA主要负责处理连续型变量 data <- gbsg[,c(-1,-3,-9,-11)] head(data) # 执行PCA pca_result <- prcomp(data, scale. = TRUE) # 使用prcomp函数进行PCA,scale. = TRUE表示对数据进行标准化处理 # 获取分析结果 get_eig(pca_result) # 绘制方差贡献图 fviz_eig(pca_result, addlabels = TRUE, ylim = c(0, 40)) # 使用fviz_eig函数绘制累计方差贡献图 # 绘制主成分贡献度图 fviz_contrib(pca_result, choice = "var", axes = 1) # 变量分别可视化 fviz_pca_var(pca_result, col.var="contrib", gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"), repel = TRUE) # 样本pca图 fviz_pca_ind(pca_result, label = "none", habillage = gbsg$age, addEllipses = TRUE )
结果展示:
> get_eig(pca_result) eigenvalue variance.percent cumulative.variance.percent Dim.1 1.8107476 25.867823 25.86782 Dim.2 1.3761590 19.659414 45.52724 Dim.3 0.9669035 13.812907 59.34014 Dim.4 0.8778691 12.540987 71.88113 Dim.5 0.8156392 11.651988 83.53312 Dim.6 0.6296778 8.995398 92.52852 Dim.7 0.5230038 7.471482 100.00000
- 「进行特征选择」
# 执行主成分分析 pca_result <- prcomp(data, scale = TRUE) # 查看主成分的方差贡献度 variance_explained <- pca_result$sdev^2 / sum(pca_result$sdev^2) # 排序主成分方差贡献度 sorted_variance <- sort(variance_explained, decreasing = TRUE) # 设置保留的主成分数量或累积方差贡献度阈值 cumulative_threshold <- 0.95 # 或根据累积方差贡献度阈值选择原始特征 cumulative_variance <- cumsum(sorted_variance) selected_features_cumulative <- data[, 1:length(which(cumulative_variance < cumulative_threshold)) + 1] # 打印选择的特征 colnames(selected_features_cumulative)
结果展示:
> colnames(selected_features_cumulative) [1] "size" "grade" "nodes" "pgr" "er" "rfstime"
过滤掉了贡献度较低的age。
- 「模型拟合」
# 拟合未处理过的逻辑回归模型 model <- glm(status ~ age + meno + size + grade + nodes + pgr + er + hormon, data = gbsg, family = binomial) # 拟合特征过滤后的逻辑回归模型 model_handle <- glm(status ~ meno + size + grade + nodes + pgr + er + hormon, data = gbsg, family = binomial) # 拟合结合主成分结果的逻辑回归 n_components <- 3 selected_features <- pca$x[, 1:n_components] data_with_pca <- cbind(gbsg, selected_features) model_pca <- glm(status ~ ., data = data_with_pca, family = binomial) # 使用逻辑回归模型进行预测 predictions <- predict(model, newdata = gbsg, type = "response") predictions_handle <- predict(model_handle, newdata = gbsg, type = "response") predictions_pca <- predict(model_pca, newdata = data_with_pca, type = "response") library(pROC) roc <- pROC::roc(gbsg$status, predictions) roc_handle <- pROC::roc(gbsg$status, predictions_handle) roc_pca <- pROC::roc(data_with_pca$status, predictions_pca) plot(roc, print.auc=TRUE, # 图像上输出AUC的值 print.auc.x=0.4, print.auc.y=0.5, # 设置AUC值坐标为(x,y) auc.polygon=TRUE, # 将ROC曲线下面积转化为多边形 auc.polygon.col="#fff7f7", # 设置ROC曲线下填充色 col="blue", # 设置ROC曲线颜色 legacy.axes=TRUE) # 使x轴从0到1,表示为1-特异度 plot.roc(roc_handle, add=TRUE, # 增加曲线 col="red", # 设置ROC曲线颜色 print.auc=TRUE, # 图像上输出AUC print.auc.x=0.4,print.auc.y=0.45) # AUC的坐标为(x,y) plot.roc(roc_pca, add=TRUE, # 增加曲线 col="green", # 设置ROC曲线颜色 print.auc=TRUE, # 图像上输出AUC print.auc.x=0.4,print.auc.y=0.4) # AUC的坐标为(x,y) legend(0.4, 0.25, # 图例位置x,y bty = "n", # 图例样式 legend=c("unhandle","feacture_selected","add_pca"), # 添加分组 col=c("blue","red","green"), # 颜色跟前面一致 lwd=2) # 线条粗细
从结果可以看出,使用PCA的结果做特征选择然后训练出的模型比不处理的要稍微差一些,但是如果把主成分分析结果作为特征参与逻辑回归,其auc有特别大的增加,大幅提升了乳腺癌的预测成功率。
五、讨论与未来展望
5.1 分析实验结果并讨论其启示和意义
通过使用PCA和逻辑回归进行乳腺癌预测,我们获得了一定的实验结果。这些结果对于乳腺癌的预测成功率提供了一些启示和意义。
首先,PCA作为一种降维技术,可以帮助我们在保持数据信息的同时减少特征的数量。使用PCA可以识别出最具有区分性的主成分,进而减少模型输入的维度。这有助于简化模型和减少模型过拟合的风险。
其次,逻辑回归作为一种分类算法,能够根据输入特征的线性组合来预测二分类输出。通过将PCA的结果作为逻辑回归模型的输入特征,我们可以利用主成分的信息来提高模型的预测性能。
实验结果表明,使用PCA和逻辑回归的组合可以提高乳腺癌预测的成功率。这意味着通过选择最具有判别性的主成分,并将其用作逻辑回归模型的输入特征,我们能够更准确地进行乳腺癌的预测。这对于早期发现和治疗乳腺癌具有重要的临床意义,可以帮助提高治疗效果和生存率。
5.2 PCA和逻辑回归的挑战和改进空间
在使用PCA和逻辑回归进行乳腺癌预测时,也存在一些挑战和改进的空间。
首先,选择主成分的数量是一个重要的问题。在实验中,我们选择了前几个具有最高方差解释比例的主成分用于逻辑回归模型。然而,如何确定最佳的主成分数量仍然是一个挑战,需要进一步的研究和优化。
其次,数据质量对于PCA和逻辑回归的结果有影响。如果数据集中存在缺失值、异常值或噪音,可能会对主成分分析和逻辑回归模型产生偏差。因此,对数据进行预处理和清洗是非常重要的,以提高模型的稳定性和预测性能。
此外,逻辑回归作为一种线性模型,对于非线性关系的建模能力有限。在未来的研究中,可以考虑使用其他更复杂的分类算法,如支持向量机或深度学习方法,以进一步提高乳腺癌预测的准确性。
5.3 未来研究方向和潜在发展前景
- 融合更多的特征:除了使用PCA选择特征,在乳腺癌预测中,可以考虑融合其他具有判别能力的特征,如基因表达数据、医学影像数据等。结合多种特征来源可以进一步提高乳腺癌预测的准确性。
- 引入领域知识:乳腺癌预测是一个复杂的问题,其中涉及大量的医学知识和专业经验。将领域知识融入模型开发过程中,可以提高模型的解释性和可靠性,进一步提高预测的准确性。
- 考虑不平衡数据集:乳腺癌数据集通常存在类别不平衡的问题,即阳性样本和阴性样本的比例不均衡。针对不平衡数据集,需要采取合适的采样策略或使用评估指标,以避免模型对多数类样本的偏好,并提高对少数类样本的预测能力。
总之,将PCA和逻辑回归应用于乳腺癌预测具有重要意义,并且有许多改进和未来发展的空间。通过进一步优化算法、改善数据质量和引入更多领域知识,我们可以提高乳腺癌预测的准确性和可靠性,为乳腺癌的早期检测和治疗提供更好的支持和指导。这对于改善患者的健康状况和生活质量具有重要的影响。
六、总结
通过本研究,我们发现使用PCA和逻辑回归的组合可以提高乳腺癌预测的成功率。具体而言,以下是我们的关键发现:
- PCA可以帮助我们识别出最具有判别性的主成分,从而减少特征的数量,并保留数据的信息。
- 选择主成分作为逻辑回归模型的输入特征,可以利用主成分的信息来提高模型的预测性能。
- 使用PCA和逻辑回归的组合可以提高乳腺癌预测的准确性和可靠性,有助于早期发现和治疗乳腺癌。
*「未经许可,不得以任何方式复制或抄袭本篇文章之部分或全部内容。版权所有,侵权必究。」