
由于之前没有接触过生物信息测序,现在刚开始运行一个校正工具遇到了一个和配置文件有关的问题,但是显示不能正常运行脚本文件,没有其他错误提示,怎么解决无从下手。。。请大佬指点迷津~
我现在用MECAT2https://github.com/xiaochuanle/MECAT2 跑一个3.4g左右大小的转录组数据,运行环境应该没有问题,因为我是在一个16g内存的阿里云服务器上做实验的,官方给的800M大的大肠杆菌基因组数据我也跑成功了,现在我按照示例 改了下运行时用的配置文件 ,但是在校正阶段就失败了,错误截图如下: 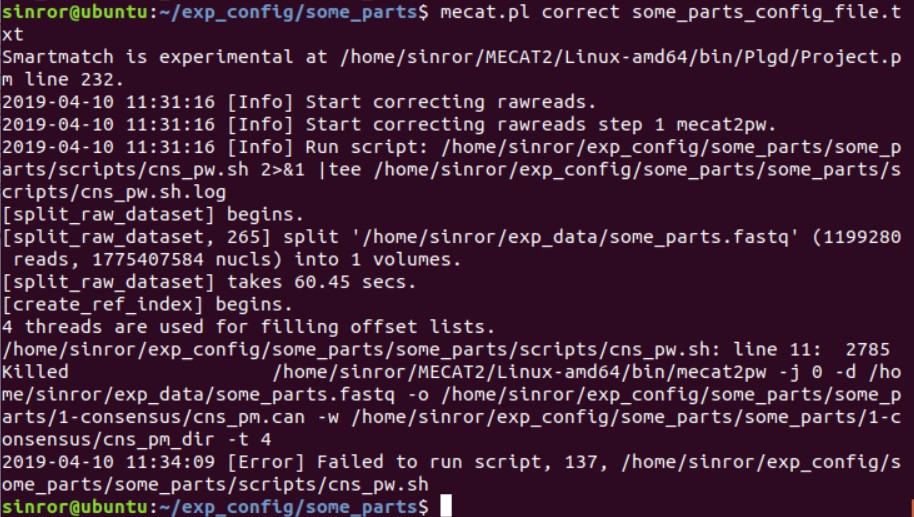 命令行中 some_parts_config_file.txt是配置文件,这个命令就是根据 配置文件内容 校正数据,结果却failed to run script,我看了这个脚本文件,只有短短几行,且和运行示例时的脚本文件一样,所以问题应该不在这个 脚本文件。
命令行中 some_parts_config_file.txt是配置文件,这个命令就是根据 配置文件内容 校正数据,结果却failed to run script,我看了这个脚本文件,只有短短几行,且和运行示例时的脚本文件一样,所以问题应该不在这个 脚本文件。
下面是我的配置文件,官网上有个模板我按照模板 填了自己的信息,但不并不确定是否填对了,所以想请 大佬们帮我看下 ,是不是因为自己的配置文件错了才运行不下去。(路径没有问题) 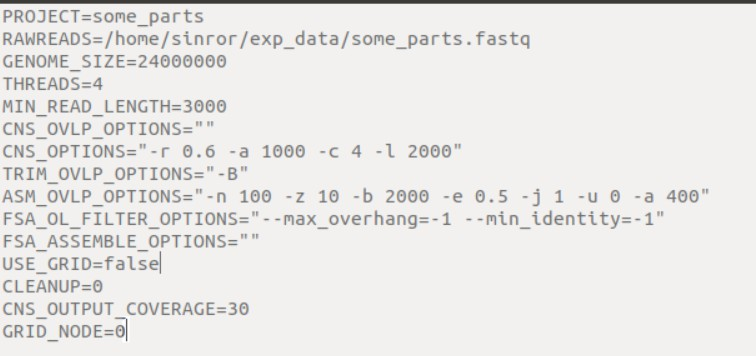 其中各字段的意义如下:从官网上截图的
其中各字段的意义如下:从官网上截图的 
遇到了同样的问题,在CSDN看到了,希望阿里云团队能够给出正确、标准的答案~请查看
版权声明:本文内容由阿里云实名注册用户自发贡献,版权归原作者所有,阿里云开发者社区不拥有其著作权,亦不承担相应法律责任。具体规则请查看《阿里云开发者社区用户服务协议》和《阿里云开发者社区知识产权保护指引》。如果您发现本社区中有涉嫌抄袭的内容,填写侵权投诉表单进行举报,一经查实,本社区将立刻删除涉嫌侵权内容。
面对您遇到的问题,首先需要确认几个关键点来定位问题所在:
配置文件格式是否正确:请仔细检查您的配置文件中是否有语法错误、字段填写是否符合要求。比如空格、引号、路径分隔符(Linux环境下通常是正斜杠/)等细节都可能导致脚本无法正确解析配置。
依赖软件版本兼容性:确保您使用的MECAT2及其依赖的软件版本与官方推荐或示例中使用的一致。不同的版本之间可能存在兼容性问题。
日志信息:虽然您提到没有其他错误提示,但通常运行失败时,程序会在标准错误输出(stderr)或者指定的日志文件中留下更详细的错误信息。请检查运行命令时是否有重定向错误输出到某个文件,如 your_command 2> error.log,然后查看这个日志文件以获取具体错误原因。
内存和资源限制:尽管您的服务器内存为16GB,但是运行大型转录组数据时,可能还需要考虑CPU使用率、磁盘I/O以及临时文件空间等因素。可以使用top或htop命令监控系统资源使用情况,确保运行过程中没有资源耗尽的情况。
权限问题:确认运行脚本的用户是否有足够的权限访问所有涉及的文件和目录。
针对您的配置文件,如果能提供具体的配置内容(注意隐去敏感信息),可能会更容易发现问题所在。不过,根据您的描述,这里有几个常见的检查点:
如果以上步骤都无法解决问题,建议直接在MECAT2的GitHub仓库中开一个Issue,附上您的配置文件(去除敏感信息)、错误日志及详细的操作步骤,项目维护者或社区成员可能会提供更针对性的帮助。同时,也可以尝试在生物信息学相关的论坛或社群(如BioStars、CSDN的生物信息板块)寻求帮助,那里有很多经验丰富的专业人士。您也可以通过ECS一键诊断全面排查并修复ECS问题。