全文链接:http://tecdat.cn/?p=6112
混合模型是k个分量分布的混合,它们共同形成混合分布:F(x )f(x)(点击文末“阅读原文”获取完整代码数据)。
F(x )= Σk = 1ķαķFķ(x )f(x)=∑k=1Kαkfk(x)
为什么要使用混合模型?
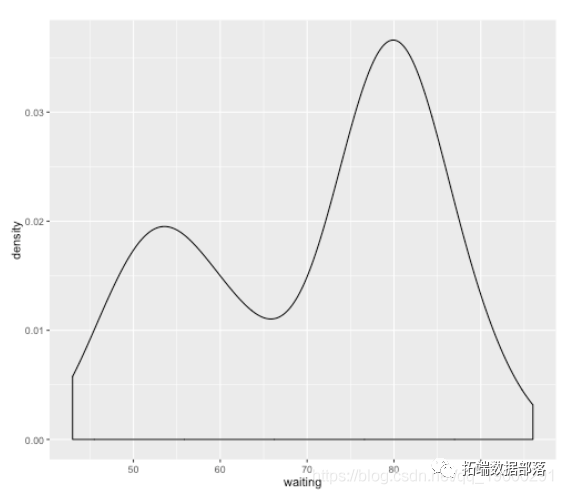
让我们通过一个例子激发您为何使用混合模型的原因。让我们说有人向您展示了以下密度图:
p <- ggplot(faithful, aes(x = waiting)) + geom_density() p
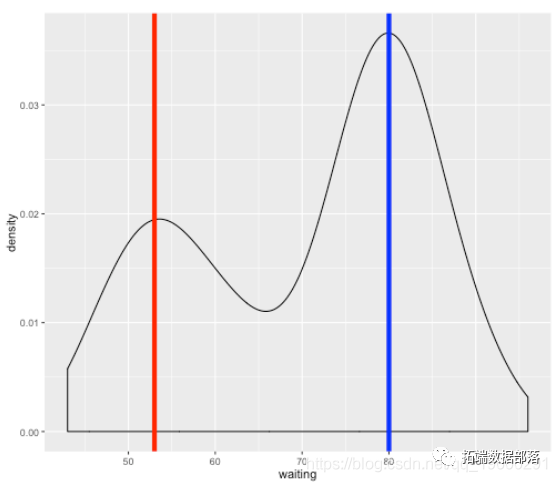
我们可以立即看到所得到的分布似乎是双峰的(即有两个凸起),表明这些数据可能来自两个不同的来源。
head(faithful) ## eruptions waiting ## 1 3.600 79 ## 2 1.800 54 ## 3 3.333 74 ## 4 2.283 62 ## 5 4.533 85 ## 6 2.883 55
该数据是2列data.frame
- 火山喷发:喷发时间(分钟)
- 等待:喷发之间的时间(分钟)
p + geom_vline(xintercept = 53, col = "red", size = 2) + geom_vline(xintercept = 80, col = "blue", size = 2)
点击标题查阅往期内容
01
02
03
04
使用高斯混合模型进行聚类
执行混合模型聚类时,您需要做的第一件事是确定要用于组件的统计分布类型。
正态分布由两个变量参数化:
- μμ
- σ2σ2
我们将用 代码来演示GMM的实际应用:
set.seed(1) wait <- faithful$waiting mixmdl <- normalmixEM(wait, k = 2)
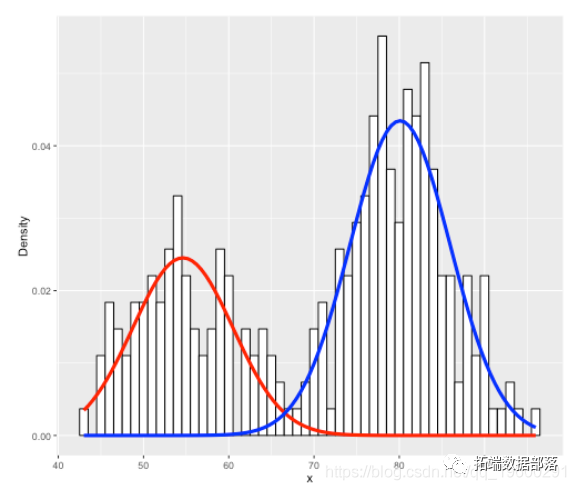
data.frame(x = mixmdl$x) %>% ggplot() + fill = "white") + stat_function(geom = "line", fun = plot_mix_comps, args = list(mixmdl$mu[1], mixmdl$sigma[1], lam = mixmdl$lambda[1]), (geom = "line", fun = plot_mix_comps, args = list(mixmdl$mu[2], mixmdl$sigma[2], lam = mixmdl$lambda[2]), colour = "blue", lwd = 1.5) + ylab("Density")
实际上很简单; 红色和蓝色线仅表示2种不同的拟合高斯分布。平均值分别为:
mixmdl$mu ## [1] 54.61489 80.09109
分别具有以下标准偏差:
mixmdl$sigma ## [1] 5.871244 5.867716
mixmdl$lambda ## [1] 0.3608869 0.6391131
另一个重要方面是每个输入数据点实际上被分配了属于这些组件之一的后验概率。我们可以使用以下代码检索这些数据:
post.df <- as.data.frame(cbind(x = mixmdl$x, mixmdl$posterior)) head(post.df, 10) #
<span style="color:#333333"><span style="color:#333333"><code>## x comp.1 comp.2 ## 1 79 0.0001030875283 0.999896912472 ## 2 54 0.9999093397312 0.000090660269 ## 3 74 0.0041357268361 0.995864273164 ## 4 62 0.9673819082244 0.032618091776 ## 5 85 0.0000012235720 0.999998776428 ## 6 55 0.9998100114503 0.000189988550 ## 7 88 0.0000001333596 0.999999866640 ## 8 85 0.0000012235720 0.999998776428 ## 9 51 0.9999901530788 0.000009846921 ## 10 85 0.0000012235720 0.999998776428 </code></span></span>
x列表示数据的值,而comp.1和comp.2分别表示属于任一组件的后验概率。
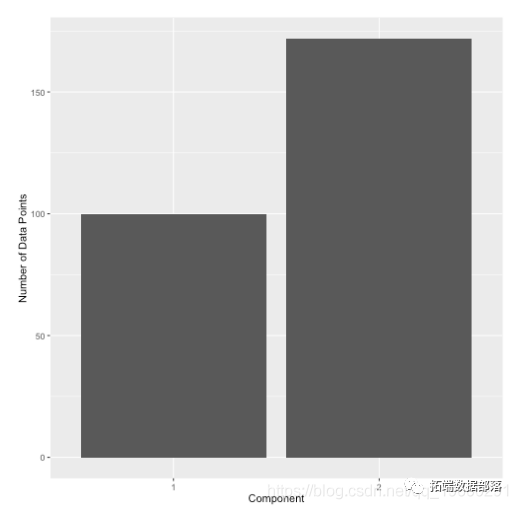
最终用户决定使用什么“阈值”将数据分配到组中。例如,可以使用0.3作为后阈值来将数据分配给comp.1并获得以下标签分布。